Biólogos constroem uma "tabela periódica" para núcleos celulares
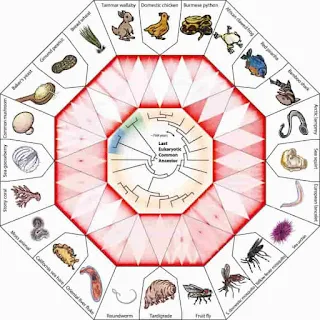
Em um artigo publicado na Science, biólogos do Baylor College of Medicine, do Netherlands Cancer Institute e da Rice University, que estudam a árvore da vida, revelam um novo sistema de classificação para núcleos celulares e a descoberta de um método para transmutar um tipo de núcleo celular em outro. Esta ilustração mostra a coleção de padrões de contato cromossômico no núcleo de vários animais e plantas. Crédito: Graphic por Adam Fotos, Olga Dudchenko, Benjamin Rowland e Erez Lieberman Aiden / Baylor College of Medicine
Cento e cinquenta anos atrás, Dmitri Mendeleev criou a tabela periódica, um sistema para classificar átomos com base nas propriedades de seus núcleos. Esta semana, uma equipe de biólogos que estuda a árvore da vida revelou um novo sistema de classificação para núcleos celulares e descobriu um método para transmutar um tipo de núcleo celular em outro.
O estudo, que aparece esta semana na revista Science, surgiu de vários esforços antes separados. Um deles centrado no DNA Zoo, um consórcio internacional que abrange dezenas de instituições, incluindo Baylor College of Medicine, o Centro de Física Teórica Biológica (CTBP) apoiado pela National Science Foundation na Rice University, na University of Western Australia e no SeaWorld.
Cientistas da equipe do DNA Zoo trabalharam juntos para classificar como os cromossomos - que podem ter vários metros de comprimento - se dobram para caber nos núcleos de diferentes espécies da árvore da vida.
“Quer estivéssemos olhando minhocas ou ouriços, ascídias ou corais, continuávamos vendo os mesmos padrões de dobra surgindo”, disse Olga Dudchenko, co-autora do novo estudo e membro do Centro de Arquitetura do Genoma em Baylor e CTBP.
Eventualmente, a equipe percebeu que estava apenas vendo variantes em dois projetos nucleares gerais. “Em algumas espécies, os cromossomos são organizados como as páginas de um jornal impresso, com as margens externas de um lado e o meio dobrado do outro”, explicou Dudchenko, que também é codiretor do DNA Zoo. “E então, em outras espécies, cada cromossomo é amassado em uma pequena bola.”
“Portanto, tínhamos um quebra-cabeça”, disse Erez Lieberman Aiden, professor associado e bolsista emérito da McNair em Baylor, codiretor do DNA Zoo e autor sênior do novo estudo. “Os dados implicam que, ao longo da evolução, as espécies podem alternar entre um tipo e outro. Nós nos perguntamos: Qual é o mecanismo de controle? Seria possível mudar um tipo de núcleo para outro no laboratório? ” Aiden também é diretor do The Center for Genome Architecture e pesquisador sênior do CTBP.
A equipe decidiu trabalhar em conjunto para confirmar o papel da condensina II. Mas então a pandemia COVID-19 atingiu, e grande parte do mundo fechou.
“Sem acesso aos nossos laboratórios, ficamos com apenas uma maneira de estabelecer o que a condensina II estava fazendo”, disse Hoencamp. “Precisávamos criar um programa de computador que pudesse simular os efeitos da condensina II na cadeia de centenas de milhões de letras genéticas que compõem cada cromossomo humano.”
A equipe recorreu a José Onuchic, a cadeira de física Harry C. e Olga K. Wiess da Rice. “Nossas simulações mostraram que, ao destruir a condensina II, você poderia fazer um núcleo humano se reorganizar para se assemelhar a um núcleo de mosca”, disse Onuchic, codiretor do CTBP, que inclui colaboradores em Rice, Baylor, Northeastern University e outras instituições em Houston e Boston .
As simulações foram realizadas por uma equipe do laboratório de Onuchic no CTBP liderada pelo bolsista de pós-doutorado e co-autora Sumitabha Brahmachari, trabalhando com Vinicius Contessoto, um ex-pós-doutorado do CTBP, e Michele Di Pierro, um investigador sênior do CTBP e atualmente um professor assistente na Universidade do Nordeste.
“Começamos com um levantamento incrivelmente amplo de dois bilhões de anos de evolução nuclear”, disse Brahmachari. “E descobrimos que muito se resume a um mecanismo simples, que podemos simular e recapitular, por conta própria, em um tubo de ensaio. É um passo empolgante no caminho para um novo tipo de engenharia de genoma - em 3D! ”



.jpg)
.jpg)
.png)

.jpg)
.jpeg)

.webp)