As cidades contêm muitas novas espécies bacterianas e virais - Atlas dos micróbios
- As cidades possuem um conjunto consistente "central" de micróbios não humanos
- Microbiomas urbanos refletem características importantes das cidades e da vida urbana
- Genes de resistência antimicrobiana são amplamente difundidos nas cidades
- As cidades contêm muitas novas espécies bacterianas e virais
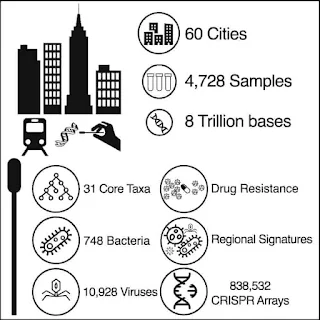
Representação gráfica do resumo do estudo, numerando o total de amostras coletadas e todos os resultados
O ambiente urbano de alta densidade historicamente abrigou apenas uma fração de todas as pessoas, com a maioria vivendo em áreas rurais ou pequenas aldeias. Nas últimas duas décadas, a situação se inverteu; 55% da população mundial agora vive em áreas urbanas (Ritchie e Roser, 2020; Nações Unidas, 2018)
As cidades também têm outras distinções características, mesmo que nunca apareçam em um cartão postal: seus micróbios residentes.
Em um período de três anos, dezenas de cientistas coletaram cerca de 5.000 amostras de 60 cidades ao redor do globo. Conforme relatado na Cell na quarta-feira (26 de maio), esses locais parecem ter comunidades microbianas distintas que incluem milhares de espécies de vírus e bactérias que nunca haviam sido documentadas antes.
“Cada cidade tem seu próprio 'eco molecular' dos micróbios que a definem”, disse o co-autor Christopher Mason
As amostras foram coletadas entre 2015 e 2017 em uma variedade de superfícies em estações de trânsito de grandes cidades. De balcões de passagem a catracas e assentos no metrô, os cientistas limpariam as superfícies por três minutos para coletar material genético para sequenciamento. Os dados, que os pesquisadores enviaram para o banco de dados de código aberto MetaSUB , mostraram que a abundância relativa de micróbios variava muito. Embora 31 espécies pudessem ser encontradas em 97 por cento das cidades - o que os autores chamam de “microbioma urbano central” - o microbioma de cada área era uma mistura única.
Desde a introdução da teoria dos germes e o trabalho de John Snow sobre a cólera, ficou claro que as pessoas nas cidades interagem com os micróbios de maneiras que podem ser marcadamente diferentes das áreas rurais (Neiderud, 2015) Micróbios no ambiente construído foram implicados como uma possível fonte de contágio (Cooley et al., 1998) e certas síndromes, como alergias, estão associadas ao aumento da urbanização (Nicolaou et al., 2005) Agora é evidente que as cidades, em geral, têm um impacto na saúde humana, embora os mecanismos desse impacto sejam amplamente variáveis e muitas vezes pouco compreendidos. Na verdade, nossa compreensão da dinâmica microbiana no ambiente urbano fora das pandemias está apenas começando (Gilbert e Stephens, 2018)
Os avanços tecnológicos no sequenciamento de última geração (NGS) e metagenômica criaram uma oportunidade sem precedentes para estudos rápidos e globais de microrganismos e seus hospedeiros, fornecendo aos pesquisadores, médicos e legisladores uma visão mais abrangente da dinâmica funcional dos microrganismos em uma cidade. NGS facilita a amostragem independente de cultura dos microrganismos em uma área com potencial para anotações taxonômicas e funcionais; isso é particularmente importante para a vigilância de microrganismos à medida que adquirem resistência antimicrobiana (AMR) (Afshinnekoo et al., 2021; Fresia et al., 2019) Os métodos metagenômicos permitem o monitoramento quase em tempo real de organismos, genes AMR e patógenos à medida que surgem dentro de uma determinada localização geográfica e têm o potencial de revelar reservatórios microbianos ocultos e detectar rotas de transmissão microbiana à medida que se espalham pelo mundo (Zhu et al., 2017) Existem vários motivadores e fontes diferentes para AMR, incluindo agricultura, agricultura e pecuária em áreas rurais e suburbanas; esgoto doméstico e industrial; uso de antimicrobianos, metais duros e biocidas; bem como dejetos humanos e animais. Todos esses fatores contribuem para a complexidade da transmissão AMR (Allen et al., 2009; Martínez, 2008; Singer et al., 2016; Thanner et al., 2016; Venter et al., 2017) Um mapa molecular de ambientes urbanos permitirá novas pesquisas significativas sobre o impacto dos microbiomas urbanos na saúde humana.
Os sistemas de transporte urbano - incluindo metrôs e ônibus - são uma interface de contato diária para bilhões de pessoas que vivem nas cidades. Os viajantes urbanos trazem consigo seus microrganismos comensais à medida que viajam e entram em contato com organismos e elementos móveis presentes no meio ambiente. O estudo do microbioma urbano e do microbioma do ambiente construído abrange vários projetos e iniciativas diferentes, incluindo trabalhos focados em sistemas de trânsito (Afshinnekoo et al., 2015; Hsu et al., 2016; Kang et al., 2018; Leung et al., 2014; MetaSUB International Consortium, 2016), hospitais (Brooks et al., 2017; Lax et al., 2017), solo (Hoch et al., 2019; Joyner et al., 2019), e esgoto (Fresia et al., 2019; Maritz et al., 2019), entre outros. Na maioria das vezes, esses esforços estudaram apenas algumas cidades selecionadas em um número limitado de ocasiões. Isso deixa uma lacuna no conhecimento científico sobre um ecossistema microbiano com o qual a população humana global interage prontamente. Microbiomas comensais humanos também foram encontrados para variar com base na cultura e, portanto, estudos isolados geograficamente são limitados e podem perder as principais diferenças (Brito et al., 2016) Além disso, dados sobre micróbios urbanos e genes AMR são urgentemente necessários nas nações em desenvolvimento, onde o consumo de drogas antimicrobianas deverá aumentar em 67% até 2030 (Nações Unidas, 2016; Van Boeckel et al., 2015), tanto das mudanças na demanda do consumidor por produtos de origem animal quanto da expansão do uso de antimicrobianos - ambos os quais podem alterar os perfis de AMR dessas cidades.
O Consórcio Internacional Metagenômica e Metadesign de Metrôs e Biomas Urbanos (MetaSUB) foi lançado em 2015 para resolver essa lacuna no conhecimento sobre a densidade, tipos e dinâmica de metagenomas urbanos e perfis AMR. Desde então, desenvolvemos protocolos de coleta e sequenciamento padronizados para processar 4.728 amostras em 60 cidades em todo o mundo ( Tabela S1 ). A amostragem ocorreu em três pontos de tempo principais: um estudo piloto em 2015-2016 e dois dias de amostragem global da cidade (21 de junho) em 2016 e 2017. Cada amostra foi sequenciada com 5-7 milhões de leituras de 125 bp pareadas usando sequenciadores NGS da Illumina ( veja Métodos STAR) Para lidar com a análise desafiadora de nosso grande conjunto de dados, geramos um pipeline de análise de código aberto (MetaSUB Core Analysis Pipeline, CAP), que inclui um conjunto abrangente de ferramentas metagenômicas de última geração revisadas por pares para taxonomia identificação, análise k-mer, predição do gene AMR, perfil funcional, de novomontagem, anotação de táxons e mapeamento geoespacial. Até onde sabemos, este estudo representa o primeiro estudo metagenômico global extenso de microbiomas urbanos. Este estudo revela um microbioma urbano “central” consistente em todas as cidades, bem como uma variação geográfica distinta que pode refletir a variação epidemiológica e que permite um novo rastreamento forense de fontes específicas para cada cidade. Nossos dados demonstram que uma fração significativa do microbioma urbano ainda precisa ser caracterizada. Embora 1.000 amostras sejam suficientes para descobrir cerca de 80% dos táxons e marcadores AMR observados, continuamos a observar táxons e genes não encontrados em outras amostras. Esta variação genética é afetada por fatores ambientais (por exemplo, clima, tipo de superfície, latitude, etc.), e as amostras mostram maior diversidade perto do equador.
As sequências associadas aos marcadores AMR são generalizadas, embora não necessariamente abundantes, e mostram especificidade geográfica. Aqui, apresentamos os resultados de nossas análises globais e um conjunto de ferramentas desenvolvidas para acessar e analisar este extenso atlas, incluindo duas visualizações interativas baseadas em mapas para amostras (metasub.org/map ) e AMRs ( resistênciaopen.org ), uma ferramenta de pesquisa indexada sobre dados de sequência bruta ( https://metagraph.ethz.ch/search ), um repositório Git para todos os pipelines analíticos e figuras, e interfaces de programação de aplicativos (APIs) para acessar resultados computacionalmente ( https://github.com/metasub/metasub_utils ).
Artigo original pdf



.jpg)
.jpeg)
.png)

.jpg)

.jpg)
